install.packages("dplyr")4 Manipulación de datos ordenados I
El paquete dplyr provee una enorme cantidad de funciones útiles para manipular y analizar datos de manera intuitiva y expresiva.
El espíritu detrás de dplyr es que la enorme mayoría de los análisis, por más complicados que sean, involucran combinar y encadenar una serie relativamente acotada de acciones (o verbos). En este curso vamos a centrarnos las cinco más comunes:
-
select(): selecciona columnas de una tabla. -
filter(): selecciona (o filtra) filas de una tabla a partir de una o más condiciones lógicas. -
group_by(): agrupa una tabla en base al valor de una o más columnas. -
mutate(): agrega nuevas columnas a una tabla. -
summarise(): calcula estadísticas para cada grupo de una tabla.
Te dieron una tabla con datos de pingüinos de 3 especies (Adelia, Barbijo y Papua). Las columnas son: especie, largo_aleta, largo_pico,
En base a esos datos, te piden que calcules la proporción promedio entre el largo de la aleta y el largo del pico para las especies Adelia y Barbijo.
¿En qué orden ejecutarías estos pasos para obtener el resultado deseado?
- usar
summarise()para calcular la estadísticamean(proporcion_aleta_pico)para cadaespecie - usar
group_by()para agrupar por la columnaespecie - usar
mutate()para agregar una columna llamadaproporcion_aleta_picocalculada comolargo_aleta/largo_pico. - usar
filter()para seleccionar las filas donde la columnaespecieno es Papua.
Para usar dplyr primero hay que instalarlo (esto hay que hacerlo una sola vez por computadora) con el comando:
y luego cargarlo en memoria con
Volvé a cargar los datos de pingüinos (para un recordatorio, podés ir a Lectura de datos ordenados):
4.1 Seleccionando columnas con select()
Para quedarse únicamente con las columnas de especie y largo del pico, usá select()
select(pinguinos, especie, largo_pico_mm)# A tibble: 344 × 2
especie largo_pico_mm
<chr> <dbl>
1 Adelia 39.1
2 Adelia 39.5
3 Adelia 40.3
4 Adelia NA
5 Adelia 36.7
6 Adelia 39.3
7 Adelia 38.9
8 Adelia 39.2
9 Adelia 34.1
10 Adelia 42
# ℹ 334 more rows¿Dónde quedó este resultado? Si te fijás en la tabla pinguinos, su formato no cambió, sigue teniendo todas las columnas originales a pesar de nuestro select:
pinguinos# A tibble: 344 × 8
especie isla largo_pico_mm alto_pico_mm largo_aleta_mm masa_corporal_g sexo
<chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
1 Adelia Torg… 39.1 18.7 181 3750 macho
2 Adelia Torg… 39.5 17.4 186 3800 hemb…
3 Adelia Torg… 40.3 18 195 3250 hemb…
4 Adelia Torg… NA NA NA NA <NA>
5 Adelia Torg… 36.7 19.3 193 3450 hemb…
6 Adelia Torg… 39.3 20.6 190 3650 macho
7 Adelia Torg… 38.9 17.8 181 3625 hemb…
8 Adelia Torg… 39.2 19.6 195 4675 macho
9 Adelia Torg… 34.1 18.1 193 3475 <NA>
10 Adelia Torg… 42 20.2 190 4250 <NA>
# ℹ 334 more rows
# ℹ 1 more variable: anio <dbl>select() y el resto de las funciones de dplyr siempre generan una nueva tabla y nunca modifican la tabla original. Para guardar la tabla con las dos columnas especie e largo_pico_mm tenés que asignar el resultado a una nueva variable.
pinguinos_isla <- select(pinguinos, especie, largo_pico_mm)
pinguinos_isla# A tibble: 344 × 2
especie largo_pico_mm
<chr> <dbl>
1 Adelia 39.1
2 Adelia 39.5
3 Adelia 40.3
4 Adelia NA
5 Adelia 36.7
6 Adelia 39.3
7 Adelia 38.9
8 Adelia 39.2
9 Adelia 34.1
10 Adelia 42
# ℹ 334 more rows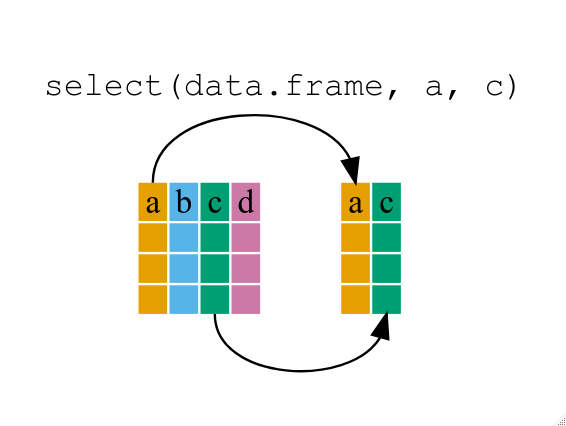
select()
4.2 Filtrando filas con filter()
Ahora podés usar filter() para quedarte con sólo unas filas. Por ejemplo, para quedarse con los pingüinos de la especie Adelia:
filter(pinguinos, especie == "Adelia")# A tibble: 152 × 8
especie isla largo_pico_mm alto_pico_mm largo_aleta_mm masa_corporal_g sexo
<chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
1 Adelia Torg… 39.1 18.7 181 3750 macho
2 Adelia Torg… 39.5 17.4 186 3800 hemb…
3 Adelia Torg… 40.3 18 195 3250 hemb…
4 Adelia Torg… NA NA NA NA <NA>
5 Adelia Torg… 36.7 19.3 193 3450 hemb…
6 Adelia Torg… 39.3 20.6 190 3650 macho
7 Adelia Torg… 38.9 17.8 181 3625 hemb…
8 Adelia Torg… 39.2 19.6 195 4675 macho
9 Adelia Torg… 34.1 18.1 193 3475 <NA>
10 Adelia Torg… 42 20.2 190 4250 <NA>
# ℹ 142 more rows
# ℹ 1 more variable: anio <dbl>La mayoría de los análisis consisten en varios pasos que van generando tablas intermedias (en el primer desafío usaste 4 pasos para calcular la proporción entre el largo de la aleta y del pico) La única tabla que te interesa es la última, por lo que ir asignando variables nuevas en cada paso intermedio es tedioso y poco práctico. Para eso se usa el operador ‘pipe’ (|>).
El operador ‘pipe’ (|>) agarra el resultado de una función y se lo pasa a la siguiente. Esto permite escribir el código como una cadena de funciones que van operando sobre el resultado de la anterior.
Las dos operaciones anteriores (seleccionar tres columnas y luego filtrar las filas correspondientes a Argentina) se pueden escribir uno después del otro y sin asignar los resultados intermedios a nuevas variables de esta forma:
# A tibble: 152 × 2
especie largo_pico_mm
<chr> <dbl>
1 Adelia 39.1
2 Adelia 39.5
3 Adelia 40.3
4 Adelia NA
5 Adelia 36.7
6 Adelia 39.3
7 Adelia 38.9
8 Adelia 39.2
9 Adelia 34.1
10 Adelia 42
# ℹ 142 more rowsLa forma de “leer” esto es “Tomá los datos en el data.frame pinguinos, después aplicale filter() tal que la especie sea Adelia, después aplicale select() para quedarte con la especie y el largo_pico_mm.
Es posible que te encuentres o te hayas encontrado con esta otra versión del operador pipe %>%. Esta es la pipe del paquete magritter, que usamos durante muchísimo tiempo (y seguimos usando) hasta que la nueva pipe |> se sumo a R 4.0.0.
Cómo vimos, el primer argumento de todas las funciones de dplyr es el data frame sobre el cual van a operar, pero notá que en las líneas con select() y filter() no escribís la tabla explícitamente.
Esto es porque la pipe implícitamente pasa el resultado de las líneas anteriores como el primer argumento de la función siguiente.
Toma el data frame pinguinos y se lo pasa al primer argumento de filter(). Luego el data frame resultante de esa operación pasa como el primer argumento de la función select() gracias al |>.
En RStudio podés escribir |> usando el atajo de teclado Ctr + Shift + M. ¡Probalo!
Completá esta cadena de código para producir una tabla que contenga los pingüinos con aletas mayores a 200 mm y la información de la especie y el largo de la aleta.
pinguinos |>
filter(largo_aleta_mm > ___) |>
select(_____, ____)Como seguramente notaste en el ejercicio cuando aplicamos filtros usamos operaciones lógicas que devuelven TRUE o FALSE según si cumplen o no con la condición. Para esto usamos operadores lógicos como ==, >, <, >=, <=, etc. R tiene varios operadores lógicos:
| Operador | Definición |
|---|---|
| < | menor que |
| x|y | x o y |
| <= | menor o igual a |
| is.na(x) | chequea si x es NA |
| !is.na(x) | chequea si x no es NA |
| > | mayor que |
| >= | mayor o igual a |
| x %in% y | chequea si x esta en y |
| !(x %in% y) | chequea si x no esta en y |
| == | igual a |
| != | no igual a |
| !x | no x |
| x & z | x y z |
Es hora de revisar lo que vimos hasta acá.
- Descargá el archivo que se encuentra en este link y guardalo en tu proyecto de trabajo en clase.
- Resolvé los ejercicios propuestos de lectura (yapa!) y de filtros. En algunos casos tendrías que reemplazar los
_____con el código correspondiente. En otros casos el ejercicio te pedirá escribir el código desde cero. - El archivo tiene más ejercicios, pero no te adelantes. Vamos a ver esos temas a continuación.
4.3 Agrupando y reduciendo con group_by() |> summarise()
Si quisieramos responder ¿cuál es el largo promedio de la aleta para cada especie de pingüino? tenemos que usar un combo de funciones: group_by() |> summarise(). Es decir, primero agrupamos las filas de nuestro data.frame según la columna especie (esto genera 3 grupos) y luego calculamos el promedio de largo_aleta_mm para cada grupo.
Vamos paso a paso.
pinguinos |>
group_by(especie) # A tibble: 344 × 8
# Groups: especie [3]
especie isla largo_pico_mm alto_pico_mm largo_aleta_mm masa_corporal_g sexo
<chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
1 Adelia Torg… 39.1 18.7 181 3750 macho
2 Adelia Torg… 39.5 17.4 186 3800 hemb…
3 Adelia Torg… 40.3 18 195 3250 hemb…
4 Adelia Torg… NA NA NA NA <NA>
5 Adelia Torg… 36.7 19.3 193 3450 hemb…
6 Adelia Torg… 39.3 20.6 190 3650 macho
7 Adelia Torg… 38.9 17.8 181 3625 hemb…
8 Adelia Torg… 39.2 19.6 195 4675 macho
9 Adelia Torg… 34.1 18.1 193 3475 <NA>
10 Adelia Torg… 42 20.2 190 4250 <NA>
# ℹ 334 more rows
# ℹ 1 more variable: anio <dbl>A primera vista parecería que la función no hizo nada, pero fijate que el resultado ahora dice que tiene grupos (“Groups:”), y nos dice qué columna es la que agrupa los datos (“especie”) y cuántos grupos hay (“3”). Cualquier operación que hagamos a continuación del group_by() van a aplicarse para cada grupo.
Para ver esto en acción, usá summarise() para computar el promedio del largo de la aleta.
# A tibble: 3 × 2
especie aleta_promedio
<chr> <dbl>
1 Adelia 190.
2 Barbijo 196.
3 Papúa 217.¡Tadá! summarise() devuelve una tabla con una columna para la especie y otra nueva, llamada “aleta_promedio” que contiene el promedio de del largo de la aleta para cada grupo.
group_by() permite agrupar en base a múltiples columnas y summarise() permite generar múltiples columnas de resumen. El siguiente código calcula la cantidad de pingüinos por especie y por isla y el promedio de su masa corporal.
pinguinos |>
group_by(especie, isla) |>
summarise(cantidad = n(),
promedio_masa_corporal = mean(masa_corporal_g, na.rm = TRUE))`summarise()` has grouped output by 'especie'. You can override using the
`.groups` argument.# A tibble: 5 × 4
# Groups: especie [3]
especie isla cantidad promedio_masa_corporal
<chr> <chr> <int> <dbl>
1 Adelia Biscoe 44 3710.
2 Adelia Dream 56 3688.
3 Adelia Torgersen 52 3706.
4 Barbijo Dream 68 3733.
5 Papúa Biscoe 124 5076.La función n() cuenta la cantidad de elementos en cada grupo, en este caso la cantidad de pingüinos por especie e isla. A diferencia de otras funciones que venimos usando no requiere de ningún argumento para funcionar en el contexto de summarise()
El resultado va a siempre ser una tabla con la misma cantidad de filas que grupos y una cantidad de columnas igual a la cantidad de columnas usadas para agrupar y los estadísticos computados.
¿Cuál te imaginás que va a ser el resultado del siguiente código? ¿Cuántas filas y columnas va a tener? (Tratá de pensarlo antes de correrlo.)
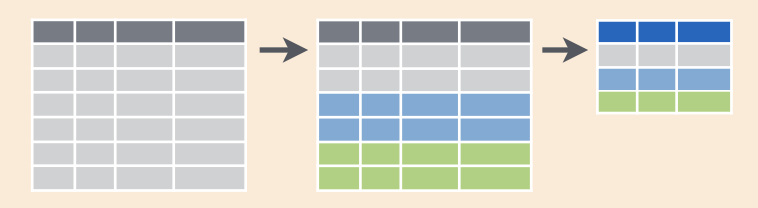
El combo group_by() |> summarise() se puede resumir en esta figura. Las filas de una tabla se dividen en grupos, y luego cada grupo se “resume” en una fila en función del estadístico usado.
4.3.1 reframe() entra a la cancha
summarise() funciona re bien hasta que que nos encontramos con la necesidad de hacer cuentas más complejas. Por ejemplo, supongamos que queremos calcular el cuantil 0.25 de la masa corporal porque decidimos que todos los pinguinos con un peso por debajo de ese valor son “pequeños”. Con lo que vimos hasta ahora podemos calcularlo.
pinguinos |>
group_by(especie) |>
summarise(cuantil_masa = quantile(masa_corporal_g, c(0.25), na.rm = TRUE))# A tibble: 3 × 2
especie cuantil_masa
<chr> <dbl>
1 Adelia 3350
2 Barbijo 3488.
3 Papúa 4700 Pero se vuelve tedioso si queremos otros cuantiles, por ejemplo el 0.75 (porque decidimos los pingüinos con masa mayor a ese valor sn “grandes”). Si intentamos extender el código anterior además del resultado vamos a terminar con un warning enorme.
pinguinos |>
group_by(especie) |>
summarise(cuantil_masa = quantile(masa_corporal_g, c(0.25, 0.75), na.rm = TRUE))Warning: Returning more (or less) than 1 row per `summarise()` group was deprecated in
dplyr 1.1.0.
ℹ Please use `reframe()` instead.
ℹ When switching from `summarise()` to `reframe()`, remember that `reframe()`
always returns an ungrouped data frame and adjust accordingly.`summarise()` has grouped output by 'especie'. You can override using the
`.groups` argument.# A tibble: 6 × 2
# Groups: especie [3]
especie cuantil_masa
<chr> <dbl>
1 Adelia 3350
2 Adelia 4000
3 Barbijo 3488.
4 Barbijo 3950
5 Papúa 4700
6 Papúa 5500 El problema es que estamos opteniendo 2 valores para cada grupo, es decir el cuantil 0.25 y el 0.75 para cada especie. En estos casos tenemos que usar reframe() que funciona de manera similar a summarise() pero está pensada para estos casos donde calculamos más de un valor por grupo.
4.4 Creando nuevas columnas con mutate()
Todo esto está bien para hacer cálculos con columnas previamente existentes, pero muchas veces tenés que crear nuevas columnas.
Podríamos querer expresar la masa corporal de los pingüinos en kilos en vez de gramos.
pinguinos |>
mutate(masa_corporal_kg = masa_corporal_g/100)# A tibble: 344 × 9
especie isla largo_pico_mm alto_pico_mm largo_aleta_mm masa_corporal_g sexo
<chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
1 Adelia Torg… 39.1 18.7 181 3750 macho
2 Adelia Torg… 39.5 17.4 186 3800 hemb…
3 Adelia Torg… 40.3 18 195 3250 hemb…
4 Adelia Torg… NA NA NA NA <NA>
5 Adelia Torg… 36.7 19.3 193 3450 hemb…
6 Adelia Torg… 39.3 20.6 190 3650 macho
7 Adelia Torg… 38.9 17.8 181 3625 hemb…
8 Adelia Torg… 39.2 19.6 195 4675 macho
9 Adelia Torg… 34.1 18.1 193 3475 <NA>
10 Adelia Torg… 42 20.2 190 4250 <NA>
# ℹ 334 more rows
# ℹ 2 more variables: anio <dbl>, masa_corporal_kg <dbl>Recordá que las funciones de dplyr nunca modifican la tabla original. mutate() devolvió una nueva tabla que es igual a la tabla pinguinos pero con la columna “masa_corporal_kg” agregada al final. Si no asignamos este resultado a una variable, no podremos usarlo luego.
La función mutate() además de permitir crear nuevas columnas, también permite modificar o actualizar columnas existentes. Para eso, solo es necesario asignarle el nombre de la columa que queremos modificar.
# A tibble: 344 × 8
especie isla largo_pico_mm alto_pico_mm largo_aleta_mm masa_corporal_g sexo
<chr> <chr> <dbl> <dbl> <dbl> <dbl> <chr>
1 ADELIA Torg… 39.1 18.7 181 3750 macho
2 ADELIA Torg… 39.5 17.4 186 3800 hemb…
3 ADELIA Torg… 40.3 18 195 3250 hemb…
4 ADELIA Torg… NA NA NA NA <NA>
5 ADELIA Torg… 36.7 19.3 193 3450 hemb…
6 ADELIA Torg… 39.3 20.6 190 3650 macho
7 ADELIA Torg… 38.9 17.8 181 3625 hemb…
8 ADELIA Torg… 39.2 19.6 195 4675 macho
9 ADELIA Torg… 34.1 18.1 193 3475 <NA>
10 ADELIA Torg… 42 20.2 190 4250 <NA>
# ℹ 334 more rows
# ℹ 1 more variable: anio <dbl>¿Te acordás del ejercicio que arrancaste cuando vimos filtros? Es hora de completarlo!
- Completá los ejercicios de este archivo que ya deberías tener en tu proyecto.
- Te vas a encontrar con ejercicios para practicar
summarise()ymutate(). De nuevo, en algunos casos tendrías que reemplazar los_____con el código correspondiente. En otros casos el ejercicio te pedirá escribir el código desde cero. - Cuando hayas terminado, knitea el archivo para generar un html y ver como queda el resultado.
4.4.1 Construyendo un paquete de R paso a paso
Ahora que ya tenés las herramientas para manipular y transformar datos ordenados, es hora de familiarizarte más con los datos de las estaciones meteorológicas.
Te proponemos responder a las siguientes preguntas manipulando los datos con los verbos de dplyr que vimos hasta ahora. Pero seguramente necesites nuevas funciones, te iremos dando pistas en el camino.
Pero antes de eso, sería útil tener todos los datos de las estaciones en un solo data.frame. Seguramente los leiste por separado y cada uno tiene un nombre distinto. La función rbind() permite unir data.frames uno debajo del otro. Usá el siguiente código y completalo para generar un data.frame con los datos de todas las estaciones.
nuevo_nombre <- rbind(___, ____, ____)Preguntas
¿Cuántas observaciones de temperatura se hicieron en cada estación?
¿Cuál es la temperatura mínima registrada y la máxima registrada en cada estación?
¿Cuál es el promedio de la temperatura de abrigo a 150 cm en cada estación? ¿Y el desvío estandar? Pista: la mayoría de las funciones tienen un argumento para sacar los
NAdel cálculo, revisá la documentación demean()y desd().¿Cuál es la proporción de
NAen temperatura de abrigo a 150 cm? Pista: podés calcular la proporción como cantidad de NA dividido la cantidad total de observaciones. La funciónis.na()devuelveTRUEsi el valor es unNA, al mismo tiempoTRUEes igual a1(así es como R lo interpreta) por lo quesum(is.na(variable))te va a dar la cantidad deNAen esa variable.¿Cuál es el promedio anual de la temperatura de abrigo a 150 cm en cada estación? Pista, podés extraer el año de la variable
fechacon anio =year(fecha)y usar esa nueva variable para agrupar los datos.¿Cuál es la precipitación acumulada mensual en cada estación? Pista: la precipitación acumulada mensual es la suma de la precipitación en cada mes. En este caso necesitas agrupar los datos por año y mes, una posibilidad es usar
floor_date(fecha, "month").